
КАТЕГОРИИ:
Архитектура-(3434)Астрономия-(809)Биология-(7483)Биотехнологии-(1457)Военное дело-(14632)Высокие технологии-(1363)География-(913)Геология-(1438)Государство-(451)Демография-(1065)Дом-(47672)Журналистика и СМИ-(912)Изобретательство-(14524)Иностранные языки-(4268)Информатика-(17799)Искусство-(1338)История-(13644)Компьютеры-(11121)Косметика-(55)Кулинария-(373)Культура-(8427)Лингвистика-(374)Литература-(1642)Маркетинг-(23702)Математика-(16968)Машиностроение-(1700)Медицина-(12668)Менеджмент-(24684)Механика-(15423)Науковедение-(506)Образование-(11852)Охрана труда-(3308)Педагогика-(5571)Полиграфия-(1312)Политика-(7869)Право-(5454)Приборостроение-(1369)Программирование-(2801)Производство-(97182)Промышленность-(8706)Психология-(18388)Религия-(3217)Связь-(10668)Сельское хозяйство-(299)Социология-(6455)Спорт-(42831)Строительство-(4793)Торговля-(5050)Транспорт-(2929)Туризм-(1568)Физика-(3942)Философия-(17015)Финансы-(26596)Химия-(22929)Экология-(12095)Экономика-(9961)Электроника-(8441)Электротехника-(4623)Энергетика-(12629)Юриспруденция-(1492)Ядерная техника-(1748)
Общая информация. Текстовая информация
|
|
|
|
Текстовая информация
Структурированная информация
Информация, хранящаяся в СИДР
Основным элементом аннотирования заболеваний в СИДР является структурированная информация, описывающая взаимодействие между двумя элементами в качестве формальной структуры субъект-предикат-объект (например, белок А повышает активность белка В). Аналогичная структура используется в таких ресурсов, как Drugbank [16] или в редакторе диаграмм процесса CellDesigner [15]. Субъекты и объекты могут представлять собой молекулы, такие как нуклеиновые кислоты, белки или химические соединения и другие элементы, такие как клеточные процессы, фенотипы или факторы внешней среды. Для того, чтобы содержание СИДРа было в стандартном формате, мы используем имена и идентификаторы из установленных ресурсов, таких как EntrezGene, KEGG [13], Чорум [24] или Менделевское наследование у человека (OMIM) [2] для аннотирования. Структурированная информация необходима для анализа взаимодействий с помощью методов биоинформатики. Тем не менее, один из нюансов этого типа данных это то, что он ограничен основной информацией эксперимента и, таким образом, не включает в себя важные детали экспериментального подхода или результатов.
Такая информация содержится в текстовой информации взаимодействий в СИДР. Это особенно полезно для ученых, проводящих эксперименты, чтобы узнать, в каких концентрациях препарат был применен в эксперименте и представляет ли он общий интерес, чтобы знать, в какой степени была увеличена экспрессия белка. Текстовая информация дает комплексное представление для объединения информации из различных источников в комплексной форме.
|
|
|
Общая информация включает в себя ссылки на литературу, соответствующую заболеванию, а также информацию, описывающую экспериментальную установку. Эта информация необходима, чтобы знать, применима ли диагностика заболевания для всех людей или же только для лиц генетически склонных к ней. Например, существует все больше доказательств того, что пол человека играет важную роль в процессах, протекающих при болезни. Для сахарного диабета 2-го типа было показано, что Gly482Ser полиморфизм PGC-1альфа (кодируемого PPARGC1A) был связан с более низким уровнем адипонектина в плазме при сахарном диабете 2 типа у мужчин, но не у женщин [21]. В неврологических заболеваниях также были обнаружены гендерные различия в протекании болезней. Дефицит XBP1 у самок мутантной линии SOD1 трансгенных мышей приводило к весьма значительному увеличению продолжительности жизни [10].
Взаимодействие данных в СИДР можно загрузить в виде плоских файлов или в Markup Language (SBML),возможен свободный и открытый обмен XML - форматами для компьютерных моделей биологических процессов [12]. Файлы в формате SBML также могут быть загружены с помощью инструментов анализа сети, таких как Cytoscape [26]. Кроме загрузки полной базы данных, СИДР также позволяет загружать взаимосвязи из результатов поиска. Графические результаты могут быть представлены в формате JPEG или в формате GraphML. Последний можно открывать и редактировать с помощью графического редактора Yed (yWorks GmbH, Тюбинген, Германия).
3 Параметры поиска и визуализация взаимодействий
Гибкий веб-интерфейc позволяет производить простой и более продвинутый поиск в СИДР. Интерфейс был разработан в тесном сотрудничестве с опытными биологами, чтобы гарантировать, что интерфейс будет интуитивно понятен и прост в использовании для конечного пользователя. Для того, чтобы обеспечить точную область поиска, СИДР предлагает его различные варианты. По умолчанию запрашиваются все взаимосвязи меж болезнями, но пользователь имеет возможность определить для поиска определенное множество заболеваний. Это особенно полезно, когда процессы при различных заболеваниях похожи. Было установлено, например, что связанные с заболеванием клеточные процессы могут влиять на развитие другой болезни, так например, связаны болезнь Альцгеймера и Диабет 2 типа. В исследованиях в Роттердаме, было показано, что сахарный диабет почти в два раза повышает риск развития деменции и болезни Альцгеймера [22].
|
|
|
Пример поиска в СИДР генов, ответственных за заболевания Alzheimer/ Parkinson/ DiabetestypeII/ Epilepsy:
1. Изучить содержание и интерфейс базы данных CIDeR database. На домашней странице данной базы в поле “Diseases”, в переводе «Заболевания» ставим галочки напротив Alzheimer/ Parkinson/ DiabetestypeII/ Epilepsy (Болезнь Альцгеймера/ Паркинсона/Диабет 2 типа/ Эпилепсия), что показано на рисунке 3.
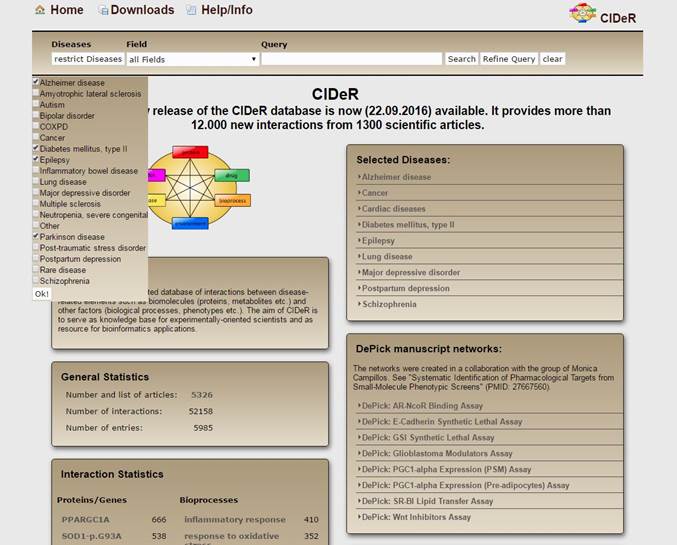
Рисунок 3 – Выбор заболеваний на домашней странице базы данных CIDeR
2. В поле “Field”, в переводе «Область», выбираем необходимый параметр поиска. Для поиска генов таких параметров будет 4, что показано на рисунке 4. Поиск может производиться по одному из 4 направлений: "Gene / Protein / Complex” («Ген/ Белок/ Соединение»), "Gene / Protein mutant / SNP” («Ген/ Мутантный белок/Однонуклеотидный полиморфизм»), "Gene / Protein variant” («Ген/ Варианты белка»), "Gene / Protein modification” («Ген/ Белковые модификации»).
Комплексный поиск и анализ вариантов позволяют более полно рассмотреть процессы, протекающие при той или иной болезни. Варианты включают в себя, среди прочего, возможность поиска, например, "Gene / Protein / Complex” («Ген/ Белок/ Соединение»), “Biological Process(GO)” («Биологический процесс»), “Tissue/ cell line (BTO)” («Ткань/ клеточная линия») или "Chemical Compound(KEGG)" («Химическое соединение»). Чтобы сделать этот процесс интуитивно понятным, все варианты перечислены в "выпадающем списке". Поиск молекул позволяет найти определенные гены или химические соединения, представляющие интерес, либо по названию, либо с помощью идентификаторов из внешних баз данных. Кроме того, СИДР хранит списки всех синонимов гена Entrez и KEGG [13]. При поиске генов или химического соединения в СИДР автоматически ищутся все возможные альтернативные названия для запроса, СИДР не ограничивается областью поиска конкретных названий или символов из определенной базы данных.
|
|
|
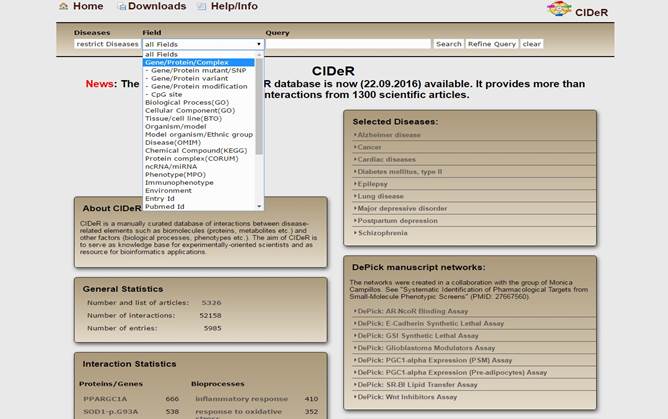
Рисунок 4 – Выбор параметров поиска на домашней странице базы данных CIDeR
3. Поле “Query”, в переводе на русский язык «Запрос», может оставаться пустым и по умолчанию в ходе поиска заполняется знаком «%», у этого поля нет выпадающего списка, и значения вбиваются вручную. После заполнения всех полей наживается кнопка “Search” («Поиск»). Поиск показывает например в %, что найдено определенное количество взаимодействий в таком-то количестве записей. Рядом с данной кнопкой имеются еще 2: “Refine query” («Детализировать запрос») и “clear” («Очистить поля»).
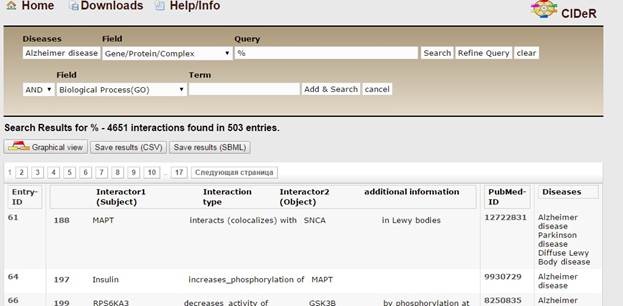 Если первоначальный поиск был закончен, он может быть дополнен последующим поиском, с использованием опции "Детализируйте запрос". Результаты второго поискового запроса связаны с первым поиском по одному из трех операторов "И", "ИЛИ" и "НЕ". Результаты поиска отображаются в виде таблицы, которая связывает с содержащимися взаимосвязями или графическим редактором, который генерирует график из результатов поиска, что показано на рисунке 5.
Если первоначальный поиск был закончен, он может быть дополнен последующим поиском, с использованием опции "Детализируйте запрос". Результаты второго поискового запроса связаны с первым поиском по одному из трех операторов "И", "ИЛИ" и "НЕ". Результаты поиска отображаются в виде таблицы, которая связывает с содержащимися взаимосвязями или графическим редактором, который генерирует график из результатов поиска, что показано на рисунке 5.
Рисунок 5 – Пример детализации запроса по болезни Альцгеймера
По каждому запросу получаем данные: “Entry-ID” («Идентификационный номер в CIDeR database»), “Interactor1(Subject)” («Субъект, который производит взаимодействия»), “Interaction type” («Тип взаимодействия»), “Interactor2(Оbject)” («Объект, на который направлены взаимодействия»), “PubMed-ID” («Ccылка на статью в PubMed, появляется при наведении на код»), “Diseases” («Заболевания в котором присутствует данное взаимодействие»). Также есть 3 кнопки над полученными данными: “Graphical view” (представление полученных данных в графическом виде, что показано на рисунке 6, однако, если получено большое количество взаимодействий, то графические данные не смогут быть отражены), “Save results (CSV)” и “Save results (SBML)”, предоставляющие возможность сохранить полученные результаты в форматах CSV и SBML.
Рисунок 6 – Графический вид всех найденных взаимодействий по болезни «Эпилепсия»
|
|
|
Все взаимосвязи заболевания могут быть интерактивно исследованы с использованием структуры yFiles (yWorks GmbH) с расширением AJAX, которая автоматически генерирует биологически правильные пути, такие как макеты сетей. На графике взаимодействия показаны в виде цветных узлов (узлами являются объекты, такие как: белок, химическое соединение или биопроцесс), которые связаны с помощью определенных отношений (ребер). График позволяет показать оптимизированную компоновку элементов, а также дает возможность реализовать интерактивные взаимодействия. При перемещении курсора мыши над краями, появляются всплывающие окна с соответствующей информацией о 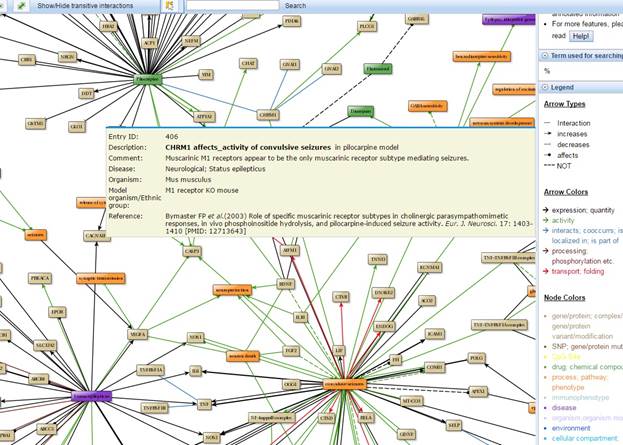 взаимодействиях, как показано на рисунке 7.
взаимодействиях, как показано на рисунке 7.
Рисунок 7 – Графический вид (приближено) всех найденных взаимодействий по болезни «Эпилепсия», показано одно из взаимодействий
Щелчок на краю открывает окно с полным аннотированным сведением об этом взаимодействии. Еще одной полезной функцией является расширение графа для интересующего элемента. Если пользователь хочет в дополнение, чтобы были показаны все взаимодействия, которые связаны с определенным узлом, ему просто нужно дважды щелкнуть мышью на элемент.
В дополнение к способности генерировать определенные пользователем графики, СИДР предоставляет предварительно определенные графики на домашней странице, где отображаются роли воспаления и митохондрий для аннотированных заболеваний и одновременно предлагается точка входа для того, чтобы изучить структуру сетей заболеваний. Здесь показаны дополнительные предопределенные графики для болезнетворных соответствующих субъектов таких, как взаимосвязь между болезнью Паркинсона и раком. Кроме того, эти страницы предлагают статистические данные о белках, химических соединениях и биологических процессах, которые прилагаются гиперссылками к соответствующему содержанию.
Целью СИДР является предоставление ресурсов знаний для ученых, ведущих экспериментальные исследования, а также представление структурированной информации для системной биологии и анализа экспериментов с высокой пропускной способностью. Углубленное руководство по аннотациям научной литературы предлагает неопытным лабораторным исследователям существенный структурированный обзор болезненных процессов. Эта информация очень подходит для создания качественных моделей и гипотез, которые будут проверены экспериментально. Особенно одновременный просмотр различных результатов в областях заболеваний, таких как неврологические заболевания, преодолевает пределы интуитивных, но неэффективных проб и ошибок.
Информация о различных заболеваниях в СИДР также позволяет выявлять потенциальных побочных эффектов лекарств, таких как побочные реакции. HSD11B1 (11-бета-гидроксистероиддегидрогеназы тип 1) катализирует превращение неактивного кортизона в активный кортизол. Открытие того, что повышенные уровни кортизола участвуют в этиологии метаболического синдрома и сахарного диабета 2 типа привело к развитию HSD11B1 ингибиторов INCB13739 и MK-0916 [8]. Так как полиморфизмы в HSD11B1 гена были связаны с повышенным риском развития болезни Альцгеймера [7] и восприимчивости к депрессии [6], ингибиторы HSD11B1 конкретно должны быть протестированы на эффективность у пациентов, страдающих болезнью Альцгеймера или депрессией.
4 Сравнение СИДР с существующими ресурсами
Одним из первых примеров, в котором отображались биологические процессы в графических сетях биохимических путей был Gerhard Michal [19]. Несмотря на несколько ресурсов, таких как Reactome [4] и PharmGKB [23], существует лишь несколько подходов, обеспечивающих сетевую структуру информации о болезни. Два известных, общедоступных примера KEGG [13] и технологические схемы, построенные с CellDesigner [14]. Концептуальный подход СИДР имеет преимущества по сравнению с информацией по заболеваниям от KEGG и CellDesigner. Во- первых, СИДР обширный, вручную курируемый ресурс, который в основном обеспечивает напрямую экспериментально доказанные взаимодействия и также связный контекст информации. Информация о болезни в KEGG также собирается с помощью ручного курирования, но в основном базируется на обзорной информации. В результате, биомедицинские информация в СИДР предлагает более подробные результаты исследований и, кроме того, представляет важную информацию, например о клеточных линиях и поле организмов. Во- вторых, в отличие от KEGG и CellDesigner, СИДР предлагает несколько вариантов поиска позволяющих искать конкретные сведения о заболевании, которые будут представлены в динамически генерируемых графиков и в качестве текстовой информации. Графическое представление в KEGG и CellDesigner статично и показывает полную информацию о болезни в одной диаграмме. Статические диаграммы полезны для презентации научно - технического прогресса в момент создания. Тем не менее, включение новых выводов в сложные диаграммы процессов, как в CellDesigner вряд ли осуществимо. В- третьих, информация о заболевании в CellDesigner и особенно KEGG ограничивается основными понятиями по данным заболеваниям. Сахарный диабет 2 типа на диаграмме в KEGG [13] состоит из 36 взаимодействий, тогда как в настоящее время в СИДР показано 6,911 взаимодействий для данного заболевания. Ограниченное количество данных в KEGG является результатом статических диаграмм, поскольку они становятся едва ли приемлемы, если должно быть показано большое количество деталей. В-четвертых, СИДР включает в себя широкий спектр заболеваний релевантных молекул, а также другие заболевания, соответствующие его факторы, биопроцессы и фенотипы. Аннотации болезней в KEGG и CellDesigner концентрируется около взаимодействия между белками. Расширение набора данных KEGG доступна из базы данных PROMISCOUS [30], которая включает взаимодействие белков лекарств из Drugbank [16]. В-пятых, информация из СИДР можно скачать не только в качестве полной базы данных, но и области, представляющей интерес, которая создается с помощью поиска. Скачать в KEGG всегда можно только полный набор данных. Пользователи, которые заинтересованы в том или ином аспекте заболевания должны разобрать соответствующие взаимодействия вручную.
Еще один ресурс, обеспеченный ручным курированием взаимодействий болезни является базой данных Сравнительная Токсикогеномика (СТ). СТ обеспечивает ручное курирование триады: химии генов, химии болезней и отношений генов болезней из литературы [5]. Для этих трех типов взаимодействий, СТ обеспечивает 260,300 точек данных, охватывающих большое разнообразие заболеваний. Концептуальное различие между этими ресурсами является то, что СТ хранит большое количество ценных взаимодействий, тогда как СИДР принимает процессно-ориентированный подход болезни и облегчает проверку взаимосвязанных сетей заболевания. Таким образом, описание патобиологических процессов для сложных заболеваний в СИДР требуется, по крайней мере, 40 различных типов взаимодействий. Следствием этого процесса-ориентированного подхода является интерактивная визуализация болезнетворных сетей, которые не доступны для СТ. Кроме того, СИДР обеспечивает аннотацию более глубоко, включая контекстную информацию, как описано выше.
Количественное сравнение показывает, что различные подходы СИДР, KEGG, СТ и CellDesigner имеют существенные различия в отношении к многообразию и абсолютному числу взаимодействий. Метаболический синдром сахарный диабет / тип 2 представляет собой расстройство, которое покрывается всеми четырьмя ресурсами. KEGG, которая охватывает широкий спектр различных биопроцессов и заболеваний, использует 36 взаимодействий и два различных типа взаимодействия (ген-ген и ген-химическое соединение), для того, чтобы аннотировать сахарный диабет 2 типа. В базе данных СТ обеспечивается в общей сложности 21,791 взаимодействий, распределенных по двум типам взаимодействия (химия -болезнь и ген-заболевание) в отношении диабета типа 2. Аннотация метаболического синдрома в формате SBML [12] с помощью диаграмм, созданных с использованием CellDesigner используют восемь различных типов взаимодействия и 97 взаимодействий. Для аннотирования диабета / метаболического синдрома у СИДР в настоящее время аннотировано 7,165 болезненных взаимодействий, которые находятся в 40 различных типах взаимодействий.
ЗАКЛЮЧЕНИЕ
В заключение хочется отметить, что большая часть других заболеваний, связанных с ресурсами посвящена конкретным болезням или специфическим молекулам. SZgene обеспечивает поле краткого обзора генетических исследований ассоциаций при шизофрении [1], dbDEPC представляет собой базу данных дифференциально экспрессированных белков в злокачественных опухолях человека [17] и MDPD представляет собой базу данных мутаций для лечения болезни Паркинсона [28]. Все эти базы данных содержат ценную информацию для исследования болезней, но они не поддерживают подходы в области системной биологии и медицины сети.
СИДР предоставляет собой общедоступный интегративный ресурс для исследователей в области неврологических и метаболических заболеваний, а также для более широкой жизни научного сообщества. Она была разработана, чтобы представить биологические факты в отображаемой сети молекулярных взаимодействий и затрагивающей пути их протекания, которые позволят ученым без особых вычислительных исследований работать с полученными данными более системно-ориентированным образом.
Список использованных источников
1. Allen NC, Bagade S, McQueen MB, Ioannidis JP, Kavvoura FK, Khoury MJ, Tanzi RE, Bertram L. Systematic meta-analyses and field synopsis of genetic association studies in schizophrenia: the SzGene database. Nat Genet. 2008;40:827–834. doi: 10.1038/ng.171.
2. Amberger J, Bocchini CA, Scott AF, Hamosh A. McKusick's Online Mendelian Inheritance in Man (OMIM). Nucleic Acids Res. 2009;37:D793–D796. doi: 10.1093/nar/gkn665.
3. Barabasi AL, Gulbahce N, Loscalzo J. Network medicine: a network-based approach to human disease. Nat Rev Genet. 2011;12:56–68. doi: 10.1038/nrg2918.
4. Croft D, O'Kelly G, Wu G, Haw R, Gillespie M, Matthews L, Caudy M, Garapati P, Gopinath G, Jassal B, Jupe S, Kalatskaya I, Mahajan S, May B, Ndegwa N, Schmidt E, Shamovsky V, Yung C, Birney E, Hermjakob H, D'Eustachio P, Stein L. Reactome: a database of reactions, pathways and biological processes. Nucleic Acids Res. 2011;39:D691–D697. doi: 10.1093/nar/gkq1018.
5. Davis AP, King BL, Mockus S, Murphy CG, Saraceni-Richards C, Rosenstein M, Wiegers T, Mattingly CJ. The Comparative Toxicogenomics Database: update 2011. Nucleic Acids Res.2011;39:D1067–D1072. doi: 10.1093/nar/gkq813.
6. Dekker MJ, Tiemeier H, Luijendijk HJ, Kuningas M, Hofman A, de Jong FH, Stewart PM, Koper JW, Lamberts SW. The effect of common genetic variation in 11beta-hydroxysteroid dehydrogenase type 1 on hypothalamic-pituitary-adrenal axis activity and incident depression. J Clin Endocrinol Metab. 2012;97:E233–E237. doi: 10.1210/jc.2011-0601.
7. De Quervain DJ, Poirier R, Wollmer MA, Grimaldi LM, Tsolaki M, Streffer JR, Hock C, Nitsch RM, Mohajeri MH, Papassotiropoulos A. Glucocorticoid-related genetic susceptibility for Alzheimer's disease. Hum Mol Genet. 2004;13:47–52.
8. Feig PU, Shah S, Hermanowski-Vosatka A, Plotkin D, Springer MS, Donahue S, Thach C, Klein EJ, Lai E, Kaufman KD. Effects of an 11beta-hydroxysteroid dehydrogenase type 1 inhibitor, MK-0916, in patients with type 2 diabetes mellitus and metabolic syndrome. Diabetes Obes Metab. 2011;13:498–504. doi: 10.1111/j.1463-1326.2011.01375.x. Rosenstock J, Banarer S, Fonseca VA, Inzucchi SE, Sun W, Yao W, Hollis G, Flores R, Levy R, Williams WV, Seckl JR, Huber R. The 11-beta-hydroxysteroid dehydrogenase type 1 inhibitor INCB13739 improves hyperglycemia in patients with type 2 diabetes inadequately controlled by metformin monotherapy. Diabetes Care. 2010;33:1516–1522. doi: 10.2337/dc09-2315.
9. Hernandez-Boussard T, Whirl-Carrillo M, Hebert JM, Gong L, Owen R, Gong M, Gor W, Liu F, Truong C, Whaley R, Woon M, Zhou T, Altman RB, Klein TE. The pharmacogenetics and pharmacogenomics knowledge base: accentuating the knowledge. Nucleic Acids Res.2008;36:D913–D918
10. Hetz C, Thielen P, Matus S, Nassif M, Court F, Kiffin R, Martinez G, Cuervo AM, Brown RH, Glimcher LH. XBP-1 deficiency in the nervous system protects against amyotrophic lateral sclerosis by increasing autophagy. Genes Dev. 2009;23:2294–2306. doi: 10.1101/gad.1830709.
11. Hoffmann R, Krallinger M, Andres E, Tamames J, Blaschke C, Valencia A. Text mining for metabolic pathways, signaling cascades, and protein networks. Sci STKE. 2005;2005:e21.
12. Hucka M, Finney A, Sauro HM, Bolouri H, Doyle JC, Kitano H, Arkin AP, Bornstein BJ, Bray D, Cornish-Bowden A, Cuellar AA, Dronov S, Gilles ED, Ginkel M, Gor V, Goryanin II, Hedley WJ, Hodgman TC, Hofmeyr JH, Hunter PJ, Juty NS, Kasberger JL, Kremling A, Kummer U, Le NN, Loew LM, Lucio D, Mendes P, Minch E, Mjolsness ED. et al. The systems biology markup language (SBML): a medium for representation and exchange of biochemical network models. Bioinformatics. 2003;19:524–531. doi: 10.1093/bioinformatics/btg015.
13. Kanehisa M, Goto S, Furumichi M, Tanabe M, Hirakawa M. KEGG for representation and analysis of molecular networks involving diseases and drugs. Nucleic Acids Res.2010;38:D355–D360. doi: 10.1093/nar/gkp896
14. Kitano H, Oda K, Kimura T, Matsuoka Y, Csete M, Doyle J, Muramatsu M. Metabolic syndrome and robustness tradeoffs. Diabetes. 2004;53(Suppl 3):S6–S15.
15. Kitano H, Funahashi A, Matsuoka Y, Oda K. Using process diagrams for the graphical representation of biological networks. Nat Biotechnol. 2005;23:961–966. doi: 10.1038/nbt1111.
16. Knox C, Law V, Jewison T, Liu P, Ly S, Frolkis A, Pon A, Banco K, Mak C, Neveu V, Djoumbou Y, Eisner R, Guo AC, Wishart DS. DrugBank 3.0: a comprehensive resource for 'omics' research on drugs. Nucleic Acids Res. 2011;39:D1035–D1041. doi: 10.1093/nar/gkq1126. Maglott D, Ostell J, Pruitt KD, Tatusova T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Res. 2011;39:D52–D57. doi: 10.1093/nar/gkq1237.
17. Li H, He Y, Ding G, Wang C, Xie L, Li Y. dbDEPC: a database of differentially expressed proteins in human cancers. Nucleic Acids Res. 2010;38:D658–D664. doi: 10.1093/nar/gkp933.
18. Mewes HW, Wachinger B, Stumpflen V. Perspectives of a systems biology of the synapse: how to transform an indefinite data space into a model?. Pharmacopsychiatry. 2010;43(Suppl 1):S2–S8.
19. Michal G. Biochemie-Atlas. Spektrum Akademischer Verlag; 1999. Biochemical pathways.
20. Najmabadi H, Hu H, Garshasbi M, Zemojtel T, Abedini SS, Chen W, Hosseini M, Behjati F, Haas S, Jamali P, Zecha A, Mohseni M, Puttmann L, Vahid LN, Jensen C, Moheb LA, Bienek M, Larti F, Mueller I, Weissmann R, Darvish H, Wrogemann K, Hadavi V, Lipkowitz B, Esmaeeli-Nieh S, Wieczorek D, Kariminejad R, Firouzabadi SG, Cohen M, Fattahi Z. et al. Deep sequencing reveals 50 novel genes for recessive cognitive disorders. Nature. 2011;478:57–63. doi: 10.1038/nature10423.
21. Okauchi Y, Iwahashi H, Okita K, Yuan M, Matsuda M, Tanaka T, Miyagawa J, Funahashi T, Horikawa Y, Shimomura I, Yamagata K. PGC-1alpha Gly482Ser polymorphism is associated with the plasma adiponectin level in type 2 diabetic men. Endocr J. 2008;55:991–997. doi: 10.1507/endocrj.K08E-070.
22. Ott A, Stolk RP, van Harskamp F, Pols HA, Hofman A, Breteler MM. Diabetes mellitus and the risk of dementia: The Rotterdam Study. Neurology. 1999;53:1937–1942. doi: 10.1212/WNL.53.9.1937.
23. Rakyan VK, Down TA, Balding DJ, Beck S. Epigenome-wide association studies for common human diseases. Nat Rev Genet. 2011;12:529–541. doi: 10.1038/nrg3000.
24. Ruepp A, Waegele B, Lechner M, Brauner B, Dunger-Kaltenbach I, Fobo G, Frishman G, Montrone C, Mewes HW. CORUM: the comprehensive resource of mammalian protein complexes - 2009. Nucleic Acids Res. 2010;38:D497–D501. doi: 10.1093/nar/gkp914.
25. Sayers EW, Barrett T, Benson DA, Bolton E, Bryant SH, Canese K, Chetvernin V, Church DM, DiCuccio M, Federhen S, Feolo M, Fingerman IM, Geer LY, Helmberg W, Kapustin Y, Landsman D, Lipman DJ, Lu Z, Madden TL, Madej T, Maglott DR, Marchler-Bauer A, Miller V, Mizrachi I, Ostell J, Panchenko A, Phan L, Pruitt KD, Schuler GD, Sequeira E. et al. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res.2011;39:D38–D51. doi: 10.1093/nar/gkq1172.
26. Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Res. 2003;13:2498–2504. doi: 10.1101/gr.1239303.
27. Su KY, Chien WL, Fu WM, Yu IS, Huang HP, Huang PH, Lin SR, Shih JY, Lin YL, Hsueh YP, Yang PC, Lin SW. Mice deficient in collapsin response mediator protein-1 exhibit impaired long-term potentiation and impaired spatial learning and memory. J Neurosci. 2007;27:2513–2524. doi: 10.1523/JNEUROSCI.4497-06.2007.
28. Tang S, Zhang Z, Kavitha G, Tan EK, Ng SK. MDPD: an integrated genetic information resource for Parkinson's disease. Nucleic Acids Res. 2009;37:D858–D862. doi: 10.1093/nar/gkn770.
29. Van Dieren S, Beulens JW, van der Schouw YT, Grobbee DE, Neal B. The global burden of diabetes and its complications: an emerging pandemic. Eur J Cardiovasc Prev Rehabil.2010;17(Suppl 1):S3–S8.
30. Von Eichborn J, Murgueitio MS, Dunkel M, Koerner S, Bourne PE, Preissner R. PROMISCUOUS: a database for network-based drug-repositioning. Nucleic Acids Res.2011;39:D1060–D1066. doi: 10.1093/nar/gkq1037.
|
|
|
|
Дата добавления: 2017-01-14; Просмотров: 158; Нарушение авторских прав?; Мы поможем в написании вашей работы!