
КАТЕГОРИИ:
Архитектура-(3434)Астрономия-(809)Биология-(7483)Биотехнологии-(1457)Военное дело-(14632)Высокие технологии-(1363)География-(913)Геология-(1438)Государство-(451)Демография-(1065)Дом-(47672)Журналистика и СМИ-(912)Изобретательство-(14524)Иностранные языки-(4268)Информатика-(17799)Искусство-(1338)История-(13644)Компьютеры-(11121)Косметика-(55)Кулинария-(373)Культура-(8427)Лингвистика-(374)Литература-(1642)Маркетинг-(23702)Математика-(16968)Машиностроение-(1700)Медицина-(12668)Менеджмент-(24684)Механика-(15423)Науковедение-(506)Образование-(11852)Охрана труда-(3308)Педагогика-(5571)Полиграфия-(1312)Политика-(7869)Право-(5454)Приборостроение-(1369)Программирование-(2801)Производство-(97182)Промышленность-(8706)Психология-(18388)Религия-(3217)Связь-(10668)Сельское хозяйство-(299)Социология-(6455)Спорт-(42831)Строительство-(4793)Торговля-(5050)Транспорт-(2929)Туризм-(1568)Физика-(3942)Философия-(17015)Финансы-(26596)Химия-(22929)Экология-(12095)Экономика-(9961)Электроника-(8441)Электротехника-(4623)Энергетика-(12629)Юриспруденция-(1492)Ядерная техника-(1748)
Цитированная. 10.5.11. Около 10% генома человека занимают два семейства транспозонов, которые, по-видимому, размножились
|
|
|
|
Общая
10-37
10.5.11. Около 10% генома человека занимают два семейства транспозонов, которые, по-видимому, размножились
лишь недавно [63]
ДНК приматов необычна по крайней мере в одном отношении: она содержит громадное количество копий двух последовательностей, про которые можно сказать, что ими прямо-таки «кишат» наши хромосомы. Оба типа этих последовательностей перемещаются в геноме в ходе РНК-
опосредованного процесса, требующего обратную транскриптазу. Одна из этих последовательностей-L1-напоминает F-элемент у дрозофилы и с.n4-
злемент у кукурузы. Полагают, что она кодирует обратную транскриптазу (см. табл. 10-3). Транспозоны обычно возникают при участии систем контроля с обратной связью, которая жестко регулирует их число в каждой клетке (и таким образом спасает клетку от возможного бедствия); тем не менее, у человека L1-элементы составляют около 4% от всей массы генома.
Еще более необычная Alu-последовательность очень коротка (около 300 нуклеотидных пар) и перемещается, встраивая копию на месте сайта-мишени. Она образовалась в результате делеции гена 7SL-PHK хозяйской клетки. Этот ген кодирует РНК-компонент сигнал-узнающей частицы (SRP-signal-recognition particle), которая принимает участие в синтезе белка. Таким образом, неясно, следует ли рассматривать Alu-
последовательность как транспозон или правильнее считать ее подвижным псевдогеном. Число копий Alu-последовательности в гаплоидном геноме человека составляет примерно 500000 (около 5% ДНК), таким образом, в среднем эта последовательность встречается один раз на каждые 5000 нуклеотидных пар ДНК. ДНК Alu транскрибируется с промотора 7SL-PHK, который узнается РНК-полимеразой III и находится внутри транскрипта. Следовательно, эта последовательность несет информацию, необходимую для своей собственной транскрипции.
|
|
|
Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К. Уотсон Дж. Д. Молекулярная биология клетки: В 3-х т. 2-е изд. перераб. и доп. Т. 2.: Пер. с англ. – М.: Мир, 1993. – 539 с.
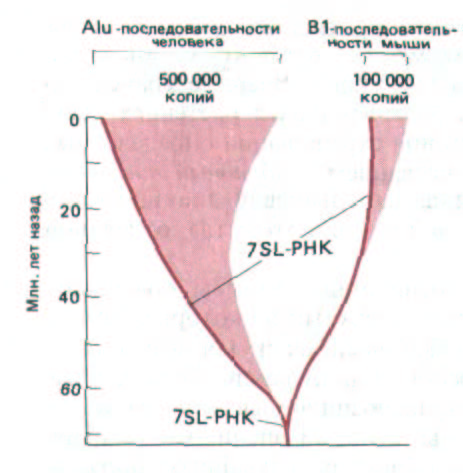
Рис. 10-74. Схема возможной эволюции повторяющихся Alu-подобных последовательностей, содержащихся в геномах мыши и человека.
Полагают, что обе эти транспозирующиеся последовательности ДНК возникли из жизненно важного гена 7SL-РНК. Однако, принимая во внимание специфическое распределение и гомологию последовательностей этих высокоповторяющичся элементов, следует признать, что увеличение их копийности происходило независимо друг от друга.
туда, куда она перемещается. Однако для перемещения Alu-последовательности необходима обратная транскршттаза.
Сравнение последовательности и расположения L1- и Alu-подобных элементов у различных млекопитающих позволяет сделать вывод, что эти элементы размножились и достигли высокой копийности относительно недавно (рис. 10-74). Трудно представить себе, что эти последовательности, рассеянные по всему геному, не оказывают заметного воздействия на близлежащие гены.
Заключение
Функциональные последовательности ДНК в геномах высших эукариот, по-видимому, собраны из небольших генетических модулей по
крайней мере двух типов. Блоки кодирующих последовательностей образуют множество комбинаций для синтеза белков; регулирующие
последовательности рассеяны среди длинных некодирующих участков и контролируют экспрессию генов. Как кодирующие последовательности
(экзоны), так и регуляторные последовательности (энхансеры) по размеру обычно не превышают нескольких сот нуклеотидных пар. В геномах
происходят разнообразные генетические рекомбинации, обусловливающие возникновение дупликацип и перенос последовательностей ДНК. В
|
|
|
некоторых случаях дуплщируются целые гены, которые могут затем приобретать новые функции. В результате рекомбинации иногда возникают
новые белки, при этом происходит перетасовка экзонов или изменение экспрессии генов за счет перекомбинации энхансеров. Перестановка
последовательностей имеет огромное значение для эволюции организмов, у эутриот она в значительной мере упрощена благодаря прерывистой
структуре генов эукариот. Важно также, что гены эукариот подвержены многочисленным активирующим и подавляющим влияниям, которые
оказывают на них разные комбинации удаленных от них энхансеров.
В геномах присутствуют различные типы транспозонов. Все вместе они составляют более 10% генома (и у дрозофилы, и у
позвоночных). Время от времени в клетках зародышевой линии происходят «транспозиционные взрывы», приводящие ко многим наследуемым
изменениям в экспрессии генов у одной и той же особи. Полагают, что транспозоны играют особую роль в эволюции, влияя на разнообразие
организмов.
Литература
Lewin В. Genes, 3rd ed. New York, Wiley, 1987.
ScHteif R, Genetics and Molecular Biology. Reading M.A., Addison-Wesley, 1986.
Stem G. S. Molecular Genetics: An Introductory Narrative. San Francisco. Freeman, 1971.
Watson J.D., Hopkins N.H., Roberts J. W., Steitz J.A., Weiner A.M. Molecular Biology of the Gene, 4th ed. Menlo Park CA, Benjamin-Cummings, 1987.
1. Gurdon J. B. The developemental capacity of nuclei taken from intestinal epithelium cells of feeding tadpoles. J. Embryol. Exp. Morphol., 10, 622-640, 1962.
Steward F. C., Mapes M. O., Mears K. Growth and organized developement of cultured cells. Am. J. Bot, 45, 705-713, 1958.
2. Garrels J, I. Changes in protein synthesis during myogenesis in a clonal cell line. Dev. Biol., 73, 134-152, 1979.
3. Darned J. K, Jr. Variety in the level of gene control in eucaryotic cells. Nature, 297, 365-371, 1982.
Derman E. et al. Transcriptional control in the production of liver-specific mRNAs. Cell, 23, 731-739, 1981.
4. Gierer A. Molecular models and combinatorial principles in cell differentiation and morphogenesis. Cold Spring Harbor Symp. Quant. Bio)., 38, 951-961, 1974.
Scott M. P., O'Fanell P. H. Spatial programming of gene expression in early Drosophila embryogenesis. Annu. Rev. Cell Biol., 2, 49-80, 1986.
Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К. Уотсон Дж. Д. Молекулярная биология клетки: В 3-х т. 2-е изд. перераб. и доп. Т. 2.: Пер. с англ. – М.: Мир, 1993. – 539 с.
5. Maniatis Т., Goodbourn S., Fischer J.A. Regulation of inducible and tissue-specific gene expression. Science, 236, 1237-1244, 1987.
Yamamoto K. Steroid receptor regulated transcription of specific genes and gene networks. Annu. Rev. Genet; 19, 209-252, 1985.
|
|
|
6. Gehring W.J., Hiromi Y. Homeotic genes and homeobox. Annu. Rev. Genet; 20, 147-173, 1986.
7. BlauH.M. et al. Plasticity of the differentiated state. Science, 230, 758-766, 1985.
Davis R. L., Weintraub H., Lassar A. B. Expression of a single transfected cDNA converts fibrpblasts to myoblasts. Cell, 51, 987-1000, 1987.
8. Miller J. H., Reznikoff W.S., eds. The Operon. Cold Spring Harbor, NY. Cold Spring Harbor Laboratory, 1978.
Neidhardt F. C. et al., eds. Escherichia coli and Salmonella typhimurium: Cellular and Molecular Biology, Vol. 2, pp. 1439-1526. Washington DC. American Society for Microbiology, 1987. (Paradigms of operon regulation in bacteria.)
Ptashne M. A. Genetic Switch. Palo Alto, CA. Blackwell, 1986.
9. Gilbert W., Muller-Hill B. The lac operator in DNA. Proc. Natl. Acad, Sci. USA, 58, 2415-2421, 1967.
Gottesman S. Bacterial regulation: global regulatory networks. Annu. Rev. Genet., 18, 415-441, 1984.
Jacob F., Monod J. Genetic regulatory mechanisms in the synthesis of proteins. J. Моl. Biol; 3, 318-356, 1961.
Reznikoff W. S., Siegele D. A., Cowing D. W., Gross C. A. The regulation of transcription initiation in bacteria. Annu. Rev. Genet., 19, 355-388, 1985.
10. de Combrugghe В., Busby S., Вис Н. Cyclic AMP receptor protein: role in transcription activation. Science, 224, 831-838, 1984.
Hochschild A., Irwin M., Ptashne M. Represser structure and the mechanism of positive control. Cell, 32, 319-325, 1983.
Raibaud O., Schwartz M. Positive control of transcription initiation in bacteria. Annu. Rev. Genet., 18, 173-206, 1984.
11. Keener J., Wong P., Popham D., Wallis J., Kustu S. A sigma factor and auxiliary protiens required for nitrogen-regulated transcription in enteric bacteria. In: RNA Polymerase and the Regulation of Transcription. (W. S. Reznikoff et al, eds.), pp. 159-175. New York, Elsevier, 1987.
Ninfa A.J., Reitzer L.J., Magasanik B. Initiation of transcription at the bacterial у/иАр2 promoter by purified E. coli components is facilitated by enhancers. Cell, 50, 1039-1046, 1987.
12. Dunn T. M., Hahn S., Ogden S., Schlief R. F. An operator at -280 base pairs that is required for the repression of araBAD operon promoter.
Proc. Natl. Acad. Sci. USA, 81, 5017-5020, 1984.
Griffith J., Hochschild A., Ptashne M. DNA loops induced by cooperative binding of lambda represser. Nature, 322, 750-752, 1986.
Mossing M. C., Record M. T. Upstream operators enhance repression of the lac promoter. Science, 233, 889-892, 1986.
13. Helmann J.D., Chamberlin M.J. Structure and function of bacterial sigma factors. Annu. Rev. Biochem; 57, 839-872, 1988.
14. Davidson B.L., Egly J.M., Mulvihill E. R., Chambon P. Formation of stable preinitiation complexes between eucaryotic class В transcription factors and promoter sequences. Nature, 301, 680-686, 1983.
Sawadogo M., Roeder R. G. Interaction of a gene-specific transcription factor with the adenovirus major late promoter upstream of the ТАТА box region. Cell, 43, 165-175, 1985.
Workman J. L., Roeder R. G. Binding of transcription factor TFIID to the major late promoter during in vitro nucleosome assembly potentiates subsequent initiation by RNA plymerase II. Cell, 51, 613-622, 1987. 15 Atchison M L. Enhancers: mechanisms of action and cell specificity.
Annu. Rev. Cell Biol: 4, 127-153, 1988.
Maniatis Т., Goodbourn S., Fischer J. Regulation of inducible and tissue-specific gene expression. Science, 236, 1237-1245, 1987.
|
|
|
McKnight S.L., Kingsbury R. Transcriptional control signals of a eucaryotic protein-coding gene. Science, 217, 316-324, 1982.
Serfling E., Jasin M., Schaffner W. Enhancers and eukaryotic gene transcription. Trends Genet., 1, 224-230, 1985.
16. Emerson B. M., Nickol J. M., Jackson P. D., Felsenfeld G. Analysis of the tissue-specific enhancer at the 3' end of the chicken adult β-globin gene. Proc. Natl. Acad. Sci. USA, 84, 4786-4790, 1987.
Evans Т., Reitman M., Felsenfeld G. An erythrocyte-specific DNA-binding factor recognizes a regulatory sequence common to all chicken globin genes. Proc. Natl. Acad. Sci. USA, 85, 5976-5980, 1988.
Jones N. C., Rigby P. W. J., Ziff E. B. Trews-acting protein factors and the regulation
Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К. Уотсон Дж. Д. Молекулярная биология клетки: В 3-х т. 2-е изд. перераб. и доп. Т. 2.: Пер. с англ. – М.: Мир, 1993. – 539 с.
of eukaryotic transcription: lessons from studies on DNA tumor viruses. Cenes Dev., 2, 267-281, 1988.
Nomiyama H., Fromental C., Xiao J. H., Chambon P. Cell-specific activity of the constituent elements of Simian virus 40 enhancer. Proc. Natl.
Acad. Sci. USA,84 7881-7885, 1987.
17. Brent R., Ptashne M. A eukaryotic transcriptional activator bearing the DNA specificity of a prokaryotic represser. Cell, 43, 729-736, 1985.
Evans R. M. The steroid and thyroid hormone receptor superfamily. Science, 240 889-895, 1988.
Kumar V. et al. Functional domains of the human estrogen receptor. Cell, 51 941-951, 1987.
Godowski P.J., Picard D., Yamamoto K. Signal transduction and transcriptionil regulation by glucocorticoid receptor - lexA fusion proteins.
Science, 241, 812-816, 1988.
18. Sen R., Baltimore D. Inducibility of kappa immunoglobulin enhancer bindingprotein NF-kappa В by a posttranslational mechanism. Cell, 47, 921-928, 1986
Yamamoto K. K., Gonzalez G.A., Biggs W.H., MontminyM.R. Phosphorylationinduced binding and transcriptional efficacy of nuclear factor CREB. Nature, 334. 494-498, 1988.
Zimarino V., Wu C. Induction of sequence-specific binding of Drosophila heat shock activator protein without pritein synthesis. Nature, 327, 727-730, 1987.
19. Metzger D., White J. H., Chambon P. The human estrogen receptor functions in yeast. Nature, 334, 31-36, 1988.
Ptashne M. Gene regulation by proteins acting nearby and at a distance. Nature, 322, 697-701, 1986. Struhl K. Promoters, activator proteins, and the mechanism of transcriptional initiation in yeast. Cell, 49, 295-297, 1987.
20. Borst P., Greaves D. R. Programmed gene rearrangements altering gene expression. Science, 235, 658-667, 1987.
Mever T. F. Molecular basis of surface antigen variation in Neisseria. Trends Genet, 3, 319-324, 1987.
Simon M., Zieg J., Silverman M., Mandel G., Doolittle R. Phase variation: evolution of a controlling element. Science, 209, 1370-1374, 1980.
21. Cross F., Hartwell L.H., Jackson C., Konopka J.B. Conjugation in Saccharomyas cerevisiae. Annu. Rev. Cell Biol; 4, 429-457, 1988.
Herskowitz I. Master regulatory loci in yeast and lambda. Cold Spring Harbor Symp. Quant. Biol., 50, 565-574, 1985.
Kushner P. J., Blair L. C., Herskowitz I. Control of yeast cell types by mobile genes: a test. Proc. Natl. Acad. Sci. USA, 76, 5264-5268, 1979.
22. Kostriken R., Strathern J. N.. Klar A., Hicks J. В., Hefforn F. A site-specific endonuclease essential for mating-type switching in Saccharomyces cerevisiae. Cell, 35, 167-174, 1983.
23. Nasmyth K., Shore D. Transcriptional regulation in the yeast life cycle. Science, 237, 1162-1170, 1987.
24. Brand A. H., Breeden L., Abraham J., Sternglanz R., Nasmyth K. Characterization of the "silencer" in yeast: a DNA sequence with properties opposite to those of a transcriptional enhancer. Cell, 41, 41-48, 1985.
25. Friedman D. I. et al. Interactions of bacteriophage and host macromolecules in the growth of bacteriophage lambda. Microbiol. Rev., 48, 299-
325, 1984.
Ptashne M. et al. How the lambda represser and его work. Cell, 19, 1-11, 1980.
26. Brown D. D. The role of stable complexes that repress and activate eukaryotic genes. Cell, 37, 359-365, 1984.
Weintraub H. Assembly and propagation of repressed and derepressed chromosomal states. Cell, 42, 705-711, 1985.
27. Brown S.W. Heterochromatin. Science, 151, 417-425, 1966.
Hsu T. C., Cooper J. E. K., Mace M. L., Brinkley B. R. Arrangement of centromeres in mouse cells. Chromosoma, 34, 73-87, 1971.
28. Gartler S. M., Riggs A. D. Mammalian X-chromosome inactivation. Annu. Rev. Genet, 17, 155-190, 1983.
Lock L. F., Takagi N., Martin G. R. Methylation of the Hprt gene on the inactive X occurs after chromosome inactivation. Cell, 48, 39-46, 1987.
Lyon M. F. X-chromosome inactivation and developmental patterns in mammals. Biol. Rev., 47, 1-35, 1972.
29. Baker W.K. Position-effect variegation. Adv. Genet, 14, 133-169, 1968.
Spofford J. B. Position-effect variegation in Drosophila. In: The Genetics and Biology of Drosophila (M. Ashburner, E. Novitski, eds.), Vol.
1C, pp. 955-1018. New York, Academic Press, 1976.
30. Goldberg D. A., Posakony J. W., Maniatis T. Correct developmental expression of a cloned alcohol dehydrogenase gene transduced into the Drosophila germ line. Cell, 34, 59-73, 1983. Grosveld F., van Assendelft G. В., Greaves D. R., Kollias G. Position independent,
Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К. Уотсон Дж. Д. Молекулярная биология клетки: В 3-х т. 2-е изд. перераб. и доп. Т. 2.: Пер. с англ. – М.: Мир, 1993. – 539 с.
high-level expression of the human β-globin gene in transgenic mice. Cell, 51, 975-985, 1987.
Meyerowitz E. M., Raghavan K. V., Mothers P. H., Roark M. How Drosophila larvae make glue: control of Sc/S-3 gene expression. Trends Genet., 3, 288-293, 1987.
Palmiter R. D., Brinster R. L. Germ-line transformation of mice. Annu. Rev. Genet., 20, 465-499, 1986.
31. Ephrussi A., Church G.M., Tonegawa S., Gilbert W. В lineage-specific interactions of an immunoglobulin enhancer with cellular factors in vivo. Science, 227, 134-140, 1985.
Garel A., Zolan M., Axel R. Genes transcribed at diverse rates have a similar conformation in chromatin. Proc. Mail. Acad. Sci. USA, 74, 4867-
4871, 1977.
Karlsson S., Nienhuis A. W. Developmental regulation of human globin genes. Annu. Rev. Biochem., 54, 1071-1108, 1985.
Weintraub H., Groudine M. Chromosomal subunits in active genes have an altered conformation. Science, 193, 848-856, 1976.
32. Brill S.J., Sternglanz R. Transcription-dependent DNA supercoiling in yeast DNA topoisomerase mutants. Cell, 54, 403-411, 1988.
Wang J.C. Superhelical DNA. Trends Biochem. Sci; 5, 219-221, 1980. Wanq ]. C., Giaever G. N. Action at a distance along a DNA. Science, 240, 300-304, 1988.
33. Guarente L. Regulatory proteins in yeast. Annu. Rev. Genet., 21, 425-452, 1987.
34. Razin A., Cedar H., Riggs A.D., eds. DNA Methylation: Biochemistry and Biological Significance. New York, Springer-Verlag, 1984.
35. Cedar H. DNA methylation and gene activity. Cell, 53, 3-4, 1988.
Ivarie R, D., Schacter B. S., O'Farrell P. H. The level of expression of the rat growth hormone gene in liver tumor cells is at least eight orders of magnitude less than that in anterior pituitary cells. Мо.. Cell. Biol., 3, 1460-1467, 1983.
Yisraeli J. et al. Muscle-specific activation of a methylated chimeric actin gene. Cell, 46, 409-416, 1986.
36. Bird A. P. CpG islands as gene markers in the vertebrate nucleus. Trends Genet, 3, 342-347, 1987.
37. Duncan L The bithorax complex. Annu. Rev. Genet; 21, 285-319, 1987.
Peifer M., Karchi F., Bender W. The bithorax complex: control of segmental identity. Genes Dev; 1, 891 -898, 1987
38. Landrick R., Yanofsky C. Transcription attenuation. In; Escherichia coli and Salmonella typhimurium: Cellular and Molecular Biology (F.C.
Neihardt et al, eds.), Vol. 2, pp. 1276 1301, Washington DC, American Society for Microbiology, 1987.
Plan T. Transcription termination and the regulation of gene expression. Annu. Rev. Biochem., 55, 339-372, 1986.
Yanofsky C. Operon-specific control by transcription attenuation. Trends Genet., 3, 356-360, 1987.
39. Andreadis A., Gallego M.E., Nadal-Ginard B. Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications. Annu. Rev. Cell Biol; 3, 207-242, 1987.
Leff S., Rosenfeld M., Evans R. Complex transcriptional units: diversity in gene expression by alternative RNA processing. Annu. Rev.
Biochem; 55, 1091-1117, 1986.
Schwarz T. L., Tempel B. L., Papazian D. M., Jan Y. N., Jan L. Y. Multiple potassium-channel components are produced by alternative splicing at the Shaker locus in Drosophila. Nature, 331, 137-142, 1988.
40. Baker B. S., Belote J. M. Sex determination and dosage compensation in Drosophila melanogaster. Annu. Rev. Genet., 17, 345-393, 1983.
Bingham P. M., Chou Т., Mims L, Zachar Z. On/off regulation of gene expression at the level of splicing. Trends Genet. 4, 134-138, 1988.
Boggs R. Т., Gregor P., Idriss S., Belote J. M., McKeown M. Regulation of sexual differentiation in Drosophila melanogaster via alternative splicing of RNA from the transformer gene. Cell, 50, 739-747, 1987.
Laski F. A., Rio D. C., RubinG.M. Tissue specificity of Drosophila P element transposition is regulated at the level of RNA splicing. Cell, 44, 7-19, 1986.
41. Early P. et al. Two mRNAs can be produced from a single immunoglobulin µ gene by alternative RNA processing pathways. Cell, 20, 313-319, 1980.
Peterson M. L., Perry R. P. Regulated production of µm and µs mRNA requires linkage of the poly(A) addition sites and is dependent on the length of the µm - µs intron. Proc. Natl. Acad. Sci. USA, 83, 8883-8887, 1986.
42. Beadle G. Genes and the chemistry of the organism. Am. Sci., 34, 31-53, 1946.
43. Newport J.W., Forbes D.J. The nucleus: structure, function, and dynamics. Annu. Rev. Biochem., 56, 535-565, 1987.
Schneider R.J., Shenk T. Impact of virus infection on host cell protein synthesis. Annu. Rev. Biochem; 56, 317-332, 1987.
44. Aziz N.; Munro H. N. Intron regulates ferritin mRNA translation through a seg-
Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К. Уотсон Дж. Д. Молекулярная биология клетки: В 3-х т. 2-е изд. перераб. и доп. Т. 2.: Пер. с англ. – М.: Мир, 1993. – 539 с.
ment of its 5' untranslated region. Proc. Natl. Acad. Sci. USA, 84, 8478-8482, 1987.
Gold L. Posttanscriptional regulatory mechanisms in Escherichia coli. Annu. Rev. Biochem., 57, 199-234, 1988.
Nomura M., Course R., Baughman G. Regulation of the synthesis of ribosomes and ribosomal components. Annu. Rev. Biochem; 53, 75-117, 1984.
Walden W. E. et al. Translational repression in eucaryotes: partial purification and characterization of a represser of ferritin in RNA translation.
Proc. Natl. Acad. Sci. USA, 85, 9503-9507, 1988.
45. Hunt T. False starts in translational control of gene expression. Nature, 316, 580-581, 1985.
Kozak M. Bifunctional messenger RNAs in eucaryotes. Cell, 47, 481-483, 1986.
Pelletier J., Sonenberg N. Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. Nature, 334, 320-325, 1988.
46. Ilan J., ed. Translational Regulation of Gene Expression. New York, Plenum Press, 1987.
Rosenthal E. Т., Hunt Т., Ruderman J. V. Selective translation of mRNA controls the pattern of protein synthesis during early development of the surf clam, Spisula solidissima. Cell, 20, 487-494, 1980.
Walden W.E., Thach R.E. Translalional control of gene expression in a normal fibroblast: characterization of a subclass of mRNAs with unusual kinetic properties. Biochemistry, 25, 2033-2041, 1986.
47. Craigen W. J., Caskey C. T. Translational frameshifting: where will it stop? Cell, 50, 1-2, 1987.
48. Casey J. L. et al. Iron-responsive elements: regulatory RNA sequences that control mRNA levels and translation. Science, 240, 924-928, 1988.
Raghow R. Regulation of messenger RNA turnover in eukaryotes. Trends Biochem. Sci., 12, 358-360, 1987.
Shaw G., Kamen R.A. conserved AU sequence from the 3' untranslated region of GM-CSF mRNA mediaters selective mRNA degradation.
Cell, 46, 659-667, 1986.
49. Graves R.A., Pandey N.B., Chodchoy N., MarzluffW.F. Translation is required for regulation of histone mRNA degradation. Cell, 48, 615-626, 1987.
MarzluffW.F., Pandey N. B. Multiple regulatory steps control histone mRNA concentrations. Trends Biochem. Sci., 13, 49-52, 1988.
Mowry K. L., Steits J. A. Identification of the human U7 snRNP as one of several factors involved in the 3' end maturation of histone premessenger RNAs. Science, 238, 1682-1687, 1987.
50. Driever W., Nusslein-Volhard C.A. gradient of bicoid protein in Drosophila embryos. Cell, 54, 83-93, 1988.
Lawrence J. В., Singer R. H. Intracellular localization of messenger RNAs for cytoskeletal protiens. Cell, 45, 407-415, 1986.
Weeks D.L., Me/ton D.A. A maternal mRNA localized to the vegetal hemisphere in Xenopus eggs codes for a growth factor related to TGFbeta.
Cell, 51, 861-867, 1987.
51. Borst P. Discontinous transcription and antigenic variation in trypanosomes. Annu. Rev. Biochem., 55, 701-732, 1986.
Eisen H. RNA editing: who's on first? Cell, 53, 331-332, 1988.
Powell L. M. et al. A novel form of tissue-specific RNA processing produces apolipoprotein-B48 in intestine. Cell, 50, 831-840, 1987. Sharp
P. A. Trans splicing: variation on a familiar theme. Cell, 50, 147-148, 1987.
52. McClain W. H., Guerrier-Takada C., Altman S. Model substrates for an RNA enzyme. Science, 238, 527-530, 1987.
Pines O., Inouye M. Antisense RNA regulation in prokaryotes. Trends Genet; 2, 284-287, 1986.
Tomizawa J. Control of ColEI plasmid replication: binding of RNA I to RNA II and inhibition of primer formation. Cell, 47, 89-97, 1986. Wu
H. N., Uhlenbeck О. С. Role of a bulged A residue in a specific RNA-protein interaction. Biochemistry, 26, 8221-8227, 1987.
53. Clarke B.C., Robertson A., Jeffreys A.J., eds. The Evolution of DNA Sequences. London. The Royal Society, 1986.
Nei M., Koehn R. K., eds. Evolution of Genes and Proteins. Sunderland MA, Sinauer, 1983.
54. Doolittle R.F., Proteins. Sci. Am; 253 (4), 88-99, 1985.
Holland S. K., Blake C. C. Proteins, exons, and molecular evolution. Biosystems, 20, 181-206, 1987.
Maeda N.. Smithies O. The evolution of multigene families: human haptoglobin gene. Annu. Rev. Genet., 20, 81-108, 1986.
55. Kourilsky P. Molecular mechanisms for gene conversion in higher cells. Trends Genet., 2, 60-63, 1986.
Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К. Уотсон Дж. Д. Молекулярная биология клетки: В 3-х т. 2-е изд. перераб. и доп. Т. 2.: Пер. с англ. – М.: Мир, 1993. – 539 с.
Roth D. В., Porter Т. N.. "Wilson J. Н. Mechanisms of uonbomologous recombination in cells. Мо.. Cell. Biol; 5, 2599-2607, 1985.
Smith G. P. Evolution of repeated DNA sequences by unequal crossovers. Science, 191, 528-535, 1976.
Stark G.R., Wahl G.M. Gene amplification. Annu. Rev. Biochem., 53, 447-491, 1984.
56. Dickerson R. E., Geis I. Hemoglobin: Structure, Function, Evolution and Pathology. Menlo Park CA, Benjamin-Cummings, 1983.
Efstratiadis A. et al. The structure and evolution of the human p-globin gene family. Cell, 21, 653-668, 1980.
Vollrath D., Nathans J., Davis R. W. Tandem array of human visual pigment genes at Xq28. Science, 240, 1669-1672, 1988.
57. Doolittle W.F. RNA mediated gene conversion? Trends Genet., 1, 64-65, 1985.
Gilbert W., Marchionni M., McKnight G. On the antiquity of introns. Cell, 46, 151-153, 1986.
Sharp P. On the origin of RNA splicing and introns. Cell, 42, 397-400, 1985.
58. Britten R.J., Kohne D.E. Repeated sequences in DNA. Science, 161, 529-540, 1968.
Jelineh W. R., Schmid C. W. Repetitive sequences in eukaryotic DNA and their expression. Annu. Rev. Biochem; 51, 813-844, 1982.
59. Craig-Holmes A. P., Shaw M. W. Polymorphism of human constitutive heterochromatin. Science, 174, 702-704, 1971.
Hsu Т. С. Human and Mammalian Cytogenetics: A Historical Perspective. New York, Springer-Verlag, 1979.
John В., Miklos G.L.G. Functional aspects of satellite DNA and heterochromatin. Int. Rev. Cytol; 58, 1-114, 1979.
Orgel L. E., Crick F. H. C. Selfish DNA: the ultimate parasite. Nature, 284, 604-607, 1980.
60. Berg D. E., Howe M. M., eds. Mobile DNA. Washington DC, American Society for Microbiology, 1989. Daring H.-P., Starlinger P. Molecular genetics of transposable elements in plants. Annu. Rev. Genet., 20, 175-200, 1986.
Finnegan D.J. Transposable elements in eukaryotes. Int. Rev. Cytol., 93, 281-326, 1985.
McCIintock B. Controlling elements and the gene. Cold Spring Harbor Symp. Quant. Biol., 21, 197-216, 1956.
61. Coen E.S., Carpenter, R. Transposable elements in Antirrhinum majus: generators of genetic diversity. Trends Genet; 2, 292-296, 1986.
Georgiev G. P. Mobile genetic elements in animal cells and their biological significance. Eur. J. Biochem., 145, 203-220, 1984.
O'Kane C.J., Gehring W. Detection in situ of genomic regulatory elements in Drosophila, Proc. Natl. Acad. Sci. USA, 84, 9123-9127, 1987.
Hiromi Y., Gehring W.J. Regulation and function of the Drosophila segmentation gene/MS/I/ tarazu. Cell, 50, 963-974, 1987.
62. Gerasimova Т.1., Mizrokhi L.J., Georgiev G.P. Transposition bursts in genetically unstable Drosophila. Nature, 309, 714-716, 1984.
McCIintock B. The significance of responses of the genome to challenge. Science, 226, 792-801, 1984.
Walbot V., Cullis C.A. Rapid genomic change in higher plants. Annu. Rev. Plant Physiol., 36, 367-396, 1985.
63. Deininger P. L., Daniels G. R. The recent evolution of mammalian repetitive DNA elements. Trands Genet; 2, 76-80, 1986.
Ruffner D. E., Sprung C. N.. Minghetti P. P., Gibbs P. E., Dugiaczyk A. Invasion of the human albumin-a-fetoprotein gene family by Alu, Kpn, and two novel repetitive DNA elements. Мо.. Biol. Evol; 4, 1-9, 1987.
Weiner A. M., Deininger P. L., Efstratiadis A. Nonviral retroposons: genes, pseudo-genes, and transposable elements generated by the reverse flow of genetic information. Annu. Rev. Biochem; 55, 631-661, 1986.
Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К. Уотсон Дж. Д. Молекулярная биология клетки: В 3-х т. 2-е изд. перераб. и доп. Т. 2.: Пер. с англ. – М.: Мир, 1993. – 539 с.
|
|
|
|
|
Дата добавления: 2015-08-31; Просмотров: 423; Нарушение авторских прав?; Мы поможем в написании вашей работы!